This is an old revision of the document!
L'explorateur de modèles biologiques
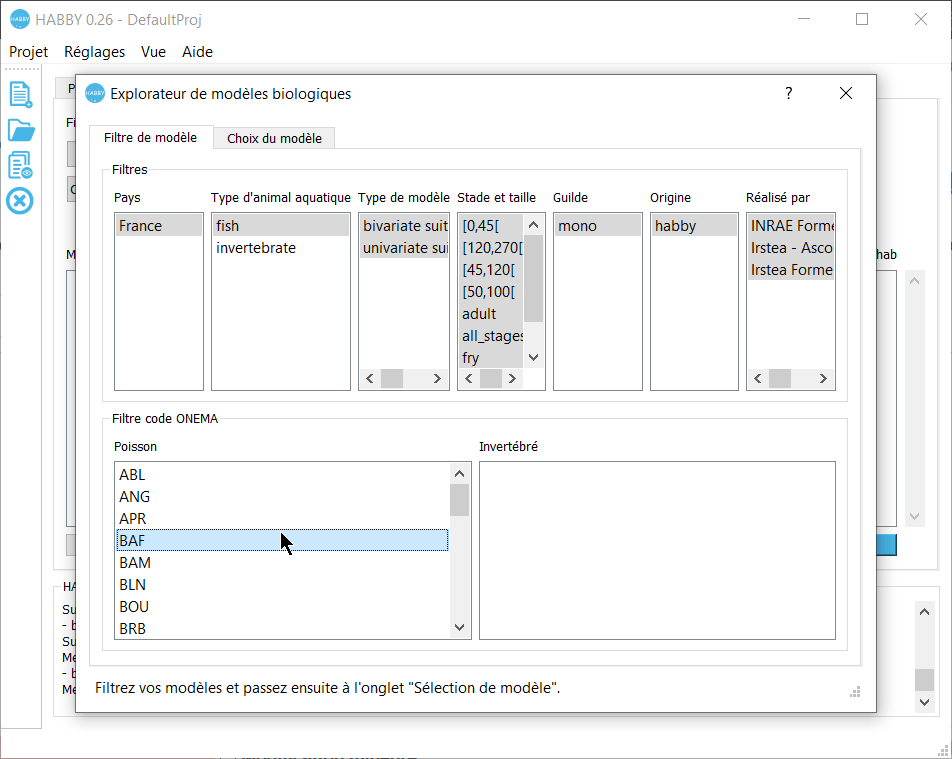
Dans ce premier onglet <hi #9BFFFF>Filtre de modèle</hi> de la fenêtre <hi #9BFFFF>Explorateur de modèle biologique</hi>, utilisez les différents <hi #9BFFFF>Filtres</hi> pour affiner votre recherche au niveau des <hi #9BFFFF>Filtres code Onema</hi>, puis passez à l'onglet suivant <hi #9BFFFF>Choix du modèle</hi>.
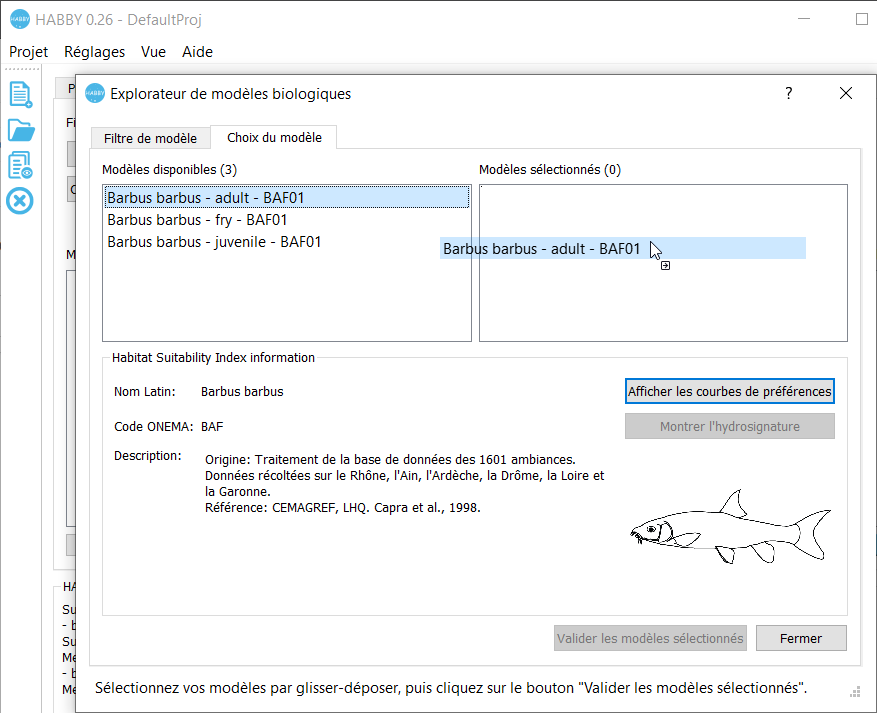
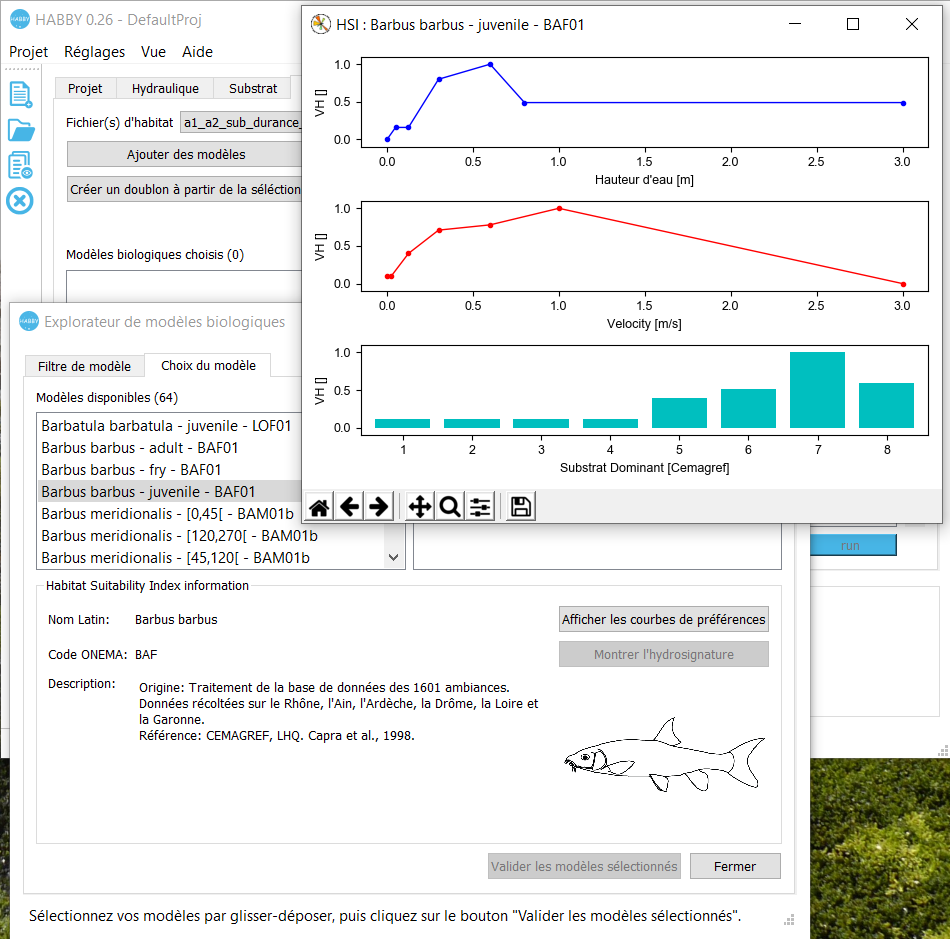
Dans ce deuxième onglet <hi #9BFFFF>Choix du modèle</hi> de la fenêtre <hi #9BFFFF>Explorateur de modèle biologique</hi>, il est possible de visualiser les modèles biologiques en graphique ainsi que leur hydrosignature quand cette dernière est disponible.
Glissez-déposez le(s) modèle(s) dans la liste de droite puis valider avec le bouton <hi #9BFFFF>Valider les modèles sélectionnés</hi> pour le(s) ajouter à l'onglet <hi #9BFFFF>Calc. hab</hi> de la fenêtre principale.
Les modèles biologiques
Généralités
Le logiciel contient une base de donnée de modèles biologiques d'un grand nombre d'espèces piscicoles.
Ajout de modèle supplémentaires
L’utilisateur à la possibilité d’ajouter ses propres modèles biologiques sous la forme de fichiers .xml dans le répertoire :
“C:\Users\USERNAME\AppData\Local\INRAE_EDF_OFB\HABBY\user_settings\biology\user_models”
Ces fichiers .xml doivent respecter la norme HABBY. Cette dernière est décrite dans le manuel de référence.